Reprodukowalność
Podczas tworzenia oprogramowania opartego o dane, niezależnie czy będą to tematy związane z analizą danych czy z ich modelowaniem (data science), jednym z podstawowych wymagań będzie reprodukowalność rozwiązań. Co ona oznacza?
Nie ma jednej definicji, ale spróbujemy sobie ten temat nieco uporządkować i pokazać jak można pójść w tym kierunku korzystając z Pythona i R.
Najpierw trochę klasyki czyli dowód istotności tematu przez cytat:
non-reproducible single occurrences are of no significance to science
Karl Popper, The Logic of Scientific Discovery
“we may say that a phenomenon is experimentally demonstrable when we know how to conduct an experiment which will rarely fail to give us statistically significant results”.
Ronald Fisher, The Design of Experiments
Czym jest reprodukowalność w nauce?
Zanim przejdziemy do danych zacznijmy od tematu reprodukowalności w badaniach naukowych. Nie wystarczy raz pokazać występowania jakiegoś zjawiska i go opisać. Wynik zostaje potwierdzony i będzie traktowany jako fakt o ile eksperyment przeprowadzony w konkretnym miejscu i czasie uda się powtórzyć gdzie indziej i kiedy indziej otrzymując ten sam wynik. Ten sam wynik oczywiście z dokładnością do losowości, co zauważył już 100 lat temu cytowany powyżej Ronald Fisher.
Ta zasada stanowi absolutna podstawa dla metody naukowej, takiej jak ją obecnie rozumiemy!
Okazuje się, że rozwój instytucjonalnej nauki, to znaczy ogromne liczby zawodowych naukowców, którzy muszą publikować regularnie nowe i ciekawe wyniki, stanowi zagrożenie dla reprodukowalności. Wynikiem oczekiwania na istotne statystycznie wyniki jest ich szukanie. A wynikiem tego szukania jest ich mniejsza istotność. Jeśli brzmi to jednocześnie zagmatwanie i ciekawie to polecam krótką, prostą i wiele tłumaczącą pracę prof. Ioannidisa z Uniwersytetu Stanforda Why Most Published Research Findings Are False.

Czym jest reprodukowalność dla przetwarzania danych?
Pewnie część czytelników wstrzymała lekturę na ostatnim paragrafie. Witamy w elitarnym klubie zainteresowanych. Jak się ma statystyczna istotność eksperymentów naukowych do Waszej codziennej pracy z danymi?
Mamy dwa poziomy reprodukowalności. Jeden bezpośrednio związany, drugi nieco luźniej.
Testy AB
Testy AB są dobrym narzędziem do podejmowania decyzji. Czy lepiej jest publikować posty w poniedziałek czy wtorek? Czy lepiej żeby strona miała tło białe czy czerwone? Czy lepiej ustawić cenę produktu na poziomie 10 złotych czy 20 złotych?
Reprodukowalność jako zdolność odtworzenia działania programu
W programach komputerowych, upraszczając, jest to odtworzenie w pełni środowiska, w którym działa program tak, żeby dla zadanych danych wejściowych dostać te same dane wyjściowe (system, biblioteki, zmienne środowiskowe, ziarno dla liczb losowych etc.). Robimy to żeby:
- Upewnić się, że nasz program będzie działać środowisku, w którym zostanie uruchomiony.
- Zdebugować kod, który zawiódł ,,produkcyjnie".
Tworząc analogię do eksperymentów naukowych, taka reprodukowalność oznacza zdolność spakowania laboratorium (razem z personelem), powielenia go, rozpakowania w dowolnym miejscu i czasie i możliwość przeprowadzenia dokładnie tego samego eksperymentu z gwarancją uzyskania dokładnie takich samych wyników.
- Reprodukowalność wzmacnia transparentność i zaufanie. Daje możliwość weryfikacji.
Kroki do osiągnięcia reprodukowalności
- Dane
- Kod
- Środowisko
Dane
Muszą być zabezpieczone (bezpieczna kopia). Koniecznie trzeba mieć dane pierwotne (raw data) i dane wejściowe do analizy (modelu). Istotne pośrednie stany, które są wynikiem czyszczenia danych również można zachować - szczególnie jeśli spodziewamy się, że będziemy do nich zaglądać w przyszłości.
Warto zadbać też o metadane. Opis zmiennych, ich typy, możliwe wartości dla zmiennych kategorycznych. W praktyce może być to np arkusz w google docs.
Kod
- Aby mieć pewność, że możemy odtworzyć analizę na datę, kiedy została ona przeprowadzona, powinniśmy korzystać z systemu kontroli wersji (Git). Zakładam, że to jest coś, co już znacie ;)
- Możliwie wysokiej jakości. Powinniśmy przestrzegać dobrych zasad tworzenia oprogramowania (więcej o tym jak to zrobić na kolejnych zajęciach)
- Wszystko powinno być zautomatyzowane tak, aby osobie, która będzie odtwrzać analizę (a możemy to być my sami za kilka tygodni czy miesięcy) będzie łatwo dokładnie odtworzyć poszczególne kroki.
Środowisko
- te same wersje pakietów
- te same zmienne środowiskowe
- te same wersje oprogramowania
- te same biblioteki systemowe
- ten sam system operacyjny
Na poprzednich zajęciach mielismy zarządzanie zależnościami, ale to jest za mało. Chcemy kontrolować całe środowisko. Ten problem rozwiązuje Docker i nim się dzisiaj zajmiemy.
Docker
Docker

Docker
- Narzędzie do uruchamianie kontenerów.
- Kontenery to pewna warstwa abstrakcji, która pozwala wykonywać zadania takie jak system operacyjny. * Oznacza to, że na dowolnym komputerze możemy uruchomić wiele różnych systemów.
- Domyślnie kontenery nie widzą siebie nawzajem (są izolowane), ale można jest ze sobą łączyć w sieci. Np. bazę danych i aplikacje z niej korzystającą.
Anatomia
- Dockerfile: lista akcji, które mają być wykonane aby utworzyć obraz dockerowy.
- Obraz dockerowy (image). System plików i zainstalowane biblioteki (zdefiniowane w Dockerfile).
- Docker container: instancja obrazu dockerowego zawierająca wszystkie niezbędne pliki do jego uruchomienia. Do kontenera możemy uruchomić, ale też się do niego podpiąć w trybie interkatywnym. Wszystkie kontenery stworzone na podstawie tego samego obrazu są identyczne.
- .dockerignore: plik zawierający ścieżki do plików, które nie mają być uwzględnione przy budowania obrazu.
Jaka jest korzyść z kontenerów?
- Na Windowsie możecie odpalić sobie wirtualnego Linuxa, na którym możecie wykonywać obliczenia tak jak na ,,prawdziwym" linuxie.
- Możecie mieć na nim zainstalowanego R w wersji 3.2 czy 4.0.
- Możecie mieć na nim dowolne wersje Pythona. Macie na nim wszystko to, czego potrzebujecie do wykonania analizy/przekształcenia/predykcji danych.
No dobrze, to jak się za to zabrać?
Najpierw tworzymy obrazy (image).
Obraz definiuje to jaki system operacyjny będziemy mieli.
Kontener to uruchomiony obraz.
Na jednym komputerze możecie mieć ten sam obraz działający w różnych kontenerach.
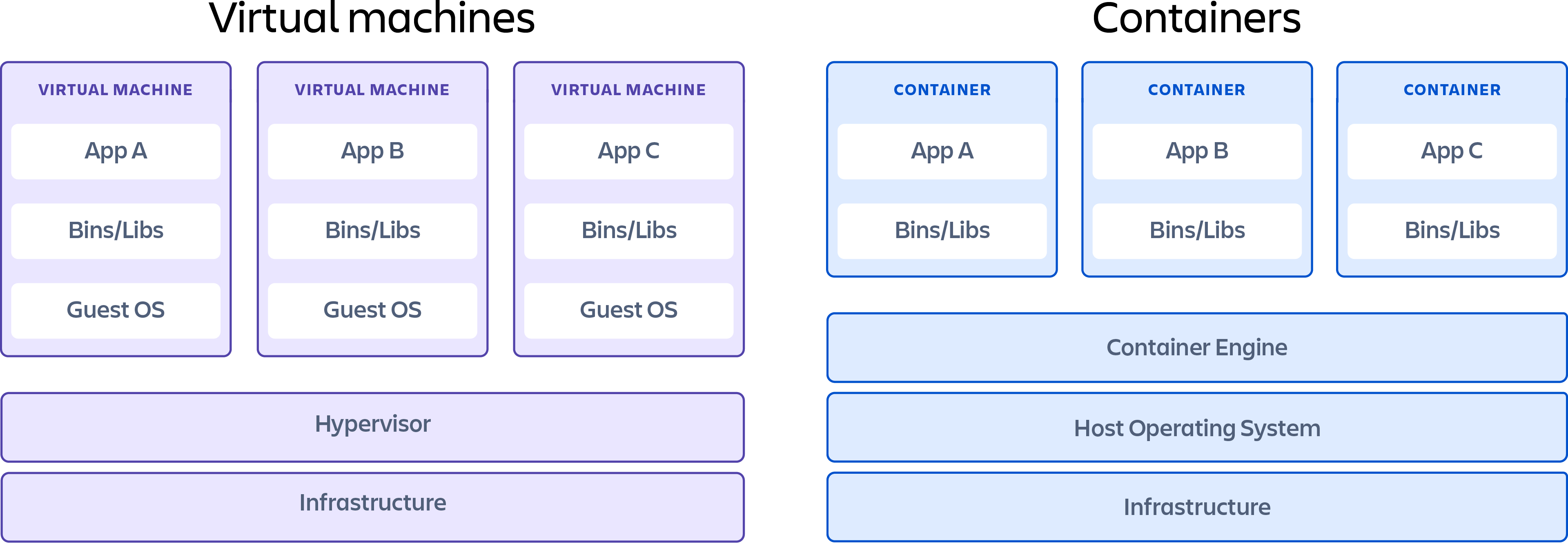
Warto zauważyć, że kontener to nie maszyna wirtualna. Różnica może wydawać się subtelna dla osoby, która dopiero zaczyna swoją przygodę z Dockerem. Konteneryzacja to rozwiązanie szybkie do tworzenia i łatwe do automatyzacji. Minusem jest potencjalnie mniejsze bezpieczeństwo. Z kolei sporą wadą (z persepktywy przetwarzania danych) przy maszynach wirtualnych (VM) jest ich interaktywny tworzenie (czyli środowisko trzeba ,,wyklikać"). Zaletą jest większe w bezpieczeństwo.
Instalacja
Zaczynamy od lokalnego zainstalowania dockera.
Oprogramowanie ściągniemy z https://docs.docker.com/get-docker/
Korzystanie z istniejących kontenerów jest darmowe.
Płatne jest umieszczanie swoich kontenerów w sieci (co ma sens, są one dosyć duże, a więc i drogie w utrzymaniu).
Można mieć własne rejestry obrazów (i wiele firm tak właśnie robi).
Pierwszy docker w R
Żeby uruchomić dockera musimy mieć zdefiniowany obraz. Albo definiujemy go sami, albo korzystamy z gotowych obrazów dostępnych on-line. Jest kilka miejsc, gdzie można je znaleźć. Dla R możemy je dostać z Rockera.
docker run --rm -ti rocker/r-base
Można podobnie uruchomić dockera, który umożliwi nam korzystanie z RStudio.
Pierwsze uruchomienia zajmuje sporo czasu, bo obraz trzeba ściągnąć z internetu. Kolejen uruchomienie będzie już szybkie.
docker run -e PASSWORD=yourpassword --rm -p 8787:8787 rocker/rstudio
Jedna linia kodu - how cool is that? Opcji jest sporo
Możemy w łatwy sposób sprawdzić jak działa nasz kod na np. najnowszej wersji Rowej konsoli lub przy jakichś konkretnych zainstalowanych bibliotekach.
Pierwszy docker w Pythonie
Podobnie jak dla R mamy bardzo wiele gotowych obrazów, z których możemy sporzystać.
docker run --rm -ti python:latest
Skąd brać obrazy?
- Podstawowy python? Zerknij tutaj.
- Notebook.
- Pytorch?.
Tworzenie własnych obrazów
Tworzenie własnych obrazów
Oczywiscie super jest odpalić RStudio w Dockerze, ale przecież chodziło nam o reprodukowalność. Więc pewnie musimy tworzyć własne obrazy? Definiujemy je w pliku Dockerfile.
Przykład, jak wygląda dockerfile:
- https://github.com/rocker-org/rocker-versioned2/blob/master/dockerfiles/r-ver_4.1.2.Dockerfile
- https://hub.docker.com/layers/python/library/python/3.9/images/sha256-8ccef93ff3c9e1bb9562d394526cdc6834033a0498073d41baa8b309f4fac20e?context=explore
Docker składa się z warstw
Kolejność ma znaczenie - docker ma warstwy.

FROM
::: incremental
- Startoway obraz.
- Może być czysty system typu ubuntu czy windows.
- Może być już przygotowanego np. obraz z działającym jupyterem lub pytorchem
- Może być coś co sami przygotowaliśmy wcześniej i jest dostępne lokalnie lub w jakimś rejestrze. :::
RUN
- Uruchom a więc każda możliwa komenda.
- Instalacja pakietów.
- Konfiguracja środowiska.
COPY
- Kopiowanie plików lokalnych do obrazu.
- W przeciwieństwie do “mount volume” to działa w jedną stronę.
- Na początku kopiujemy tylko niezbedne pliki typu
requirements.txt. - Cała reszta (niemal) na samym końcu.
CMD
- Uruchomienie komendy, która ma zostać wykonana w Dockerze.
- Wszystkie poprzednie kroki mają sprawić, że ta komenda wykona się poprawnie.
Budowanie obrazu
docker build -t nameofdockerimage .
-t tag, tagging image--build-arg IMAGE_REGISTRY=http://url_here.com, przekazanie argumentu do budowanego obrazu-f pathto/Dockerfile, non-default Dockerfile
Uruchomienie kontenera
docker run --rm nameofdockerimage
-it, interaktywne.-v full_path_local_data:/data/, montowanie folderu.-p, forwardowanie portów np.-p 8788:8787.-d,--detach, kontener będzie odpalony w tle.
Współdzielone foldery
- Przy uruchomieniu kontenera istnieje możliwość udostępnienia folderu, który będzie współdzielony między kontenerem a dyskiem na maszynie.
- Flaga
-vlub--volume. - Np.
-v /Users/bob/Documents:/home/rstudio/Documents rocker/rstudio. Ważne, że ścieżki są absolute nie relative.
docker run -d -e PASSWORD=yourpassword -p 8787:8787
-v /Users/bob/Documents:/home/rstudio/Documents rocker/rstudio
Zmienne środowiskowe
::: incremental
- Popularny sposób przekazania konfiguracji do kontenera.
--env-file ./env.list, przekazanie pliku zawierającego zmienne środowiskowe.-e PASSWORD=yourpassword, przekazanie explicite jednej konkretnej zmiennej. :::
Zarządzanie obrazami
docker system prune- usuwa wszystkie zasoby (images, containers, volumes, and networks) które nie są otagowane lub powiązane z jakimś kontenerem (dangling).docker system prune -ausuwa dodatkowo wszystkie zatrzymane kontenery i nieużywane obrazy.docker images -adocker rmi IMAGE_NAME/IMAGE_IDdocker images -a | grep "pattern" | awk '{print $3}' | xargs docker rmi- usuwanie wszystkich obrazów zawierających pattern.
Zarządzanie kontenerami
::: incremental
- Kontenery także nie są małe - warto zadbać o porządek!
docker ps -a- wylistuje nam chodzące i zatrzymane kontenery.docker run --rm- usuwamy kontener po jego zastopowaniu.docker rm -v $(docker ps -a -q), usuwamy wszystkie zastopowane kontenery. :::
Demo
Uwagi do demo
- https://github.com/psobczyk/dash-demo
- Proszę zwrócić uwagę, że na Windowsie adres 0.0.0.0 nie jest automatycznie przekierowany na localhosta. Trzeba więc zmodyfikować adres jaki podaje gunicorn.
- Nasze
requirements.inpowiększyło się o pakietygunicorniredis.
Gdzie przechowujemy obrazy?
- Przechowywanie obrazów jest płatne, bo obrazy są duże.
- Dockerhub, Github container registry, Gitlab registry, AWS ECR etc. etc.
- Własny rejestr.
Docker compose
Imago solus, nunquam solus
Sam obraz nie wystarcza - ważne jest całe środowisko.
Nasza aplikacja może potrzebować innych usług np. bazy danych, redis-a etc.
Docker compose tworzy całe środowisko. Kilka usług będzie odpalonych w tej samej sieci i będą one w stanie się między sobą komunikować.
Przykład z internetu
version: '3'
services:
myapp:
build:
.
depends_on:
- "myapp-redis"
ports:
- "8000:8000"
# redis cache for storing data
myapp-redis:
image: redis
expose:
- "6379"
Demo
https://github.com/psobczyk/dash-demo/blob/master/docker-compose.yml
Żródła
- https://towardsdatascience.com/docker-for-data-scientists-part-1-41b0725d4a50
- https://towardsdatascience.com/dockerize-your-dash-app-1e155dd1cea3
- https://dagshub.com/blog/setting-up-data-science-workspace-with-docker/
- https://towardsdatascience.com/deploy-your-ai-engine-in-any-other-machine-using-docker-docker-compose-1691e57cb866
- https://esajournals.onlinelibrary.wiley.com/doi/full/10.1002/bes2.1801